上海师范大学黄学辉团队开发水稻QTL基因变异图谱导航系统
- 分类:新闻动态
- 作者:
- 来源:
- 发布时间:2021-02-02 17:08
- 访问量:
【概要描述】北京时间2021年2月2日凌晨0时,上海师范大学黄学辉团队在Nature Genetics发表了题为“A quantitative genomics map of rice provides genetic insights and guides breeding”的封面文章。 该研究构建了迄今为止最完善的水稻数量性状基因关键变异(causative variation)图谱,开发了一款智能化的水稻育种导航程序。
上海师范大学黄学辉团队开发水稻QTL基因变异图谱导航系统
【概要描述】北京时间2021年2月2日凌晨0时,上海师范大学黄学辉团队在Nature Genetics发表了题为“A quantitative genomics map of rice provides genetic insights and guides breeding”的封面文章。
该研究构建了迄今为止最完善的水稻数量性状基因关键变异(causative variation)图谱,开发了一款智能化的水稻育种导航程序。
- 分类:新闻动态
- 作者:
- 来源:
- 发布时间:2021-02-02 17:08
- 访问量:
北京时间2021年2月2日凌晨0时,上海师范大学黄学辉团队在Nature Genetics发表了题为“A quantitative genomics map of rice provides genetic insights and guides breeding”的封面文章。
该研究构建了迄今为止最完善的水稻数量性状基因关键变异(causative variation)图谱,开发了一款智能化的水稻育种导航程序。
该研究为水稻遗传研究提供了全面的数量性状基因信息,建立了水稻分子设计育种新方法,有望为水稻新品种的快速培育提供技术支持。

基因关键变异的电子图谱
最近的二十年里,大量的水稻QTL基因被克隆。但是,这些基因的关键等位变异信息尚未被精准、系统地梳理过。论文作者利用Web of Science数据库检索,从大量水稻基因文献中,剔除了来自突变体定位或反向遗传学研究的基因,通过逐一阅读核对,确定了225个已报道的水稻QTL基因。
进一步,结合水稻基因组序列和文献中等位变异的图示或描述,将关键功能变异位点(QTN)逐一锚定到水稻基因组精确的位置上,最终获得一张包含348个变异位点和562个等位基因的分子图谱(QTN map)。这些关键变异中76.1%位于编码区,其余主要位于上游调控区。在这225个基因中,有56个基因存在复等位,含有多个QTN。
研究人员根据水稻QTN图谱,收集了来自26个国家的404份种质材料,构建了包含各类稀有等位基因的实体库(根据全基因组测序和QTN鉴定,覆盖了电子图谱中562个等位基因的95.5%),为水稻遗传改良配备了丰富的供体资源。
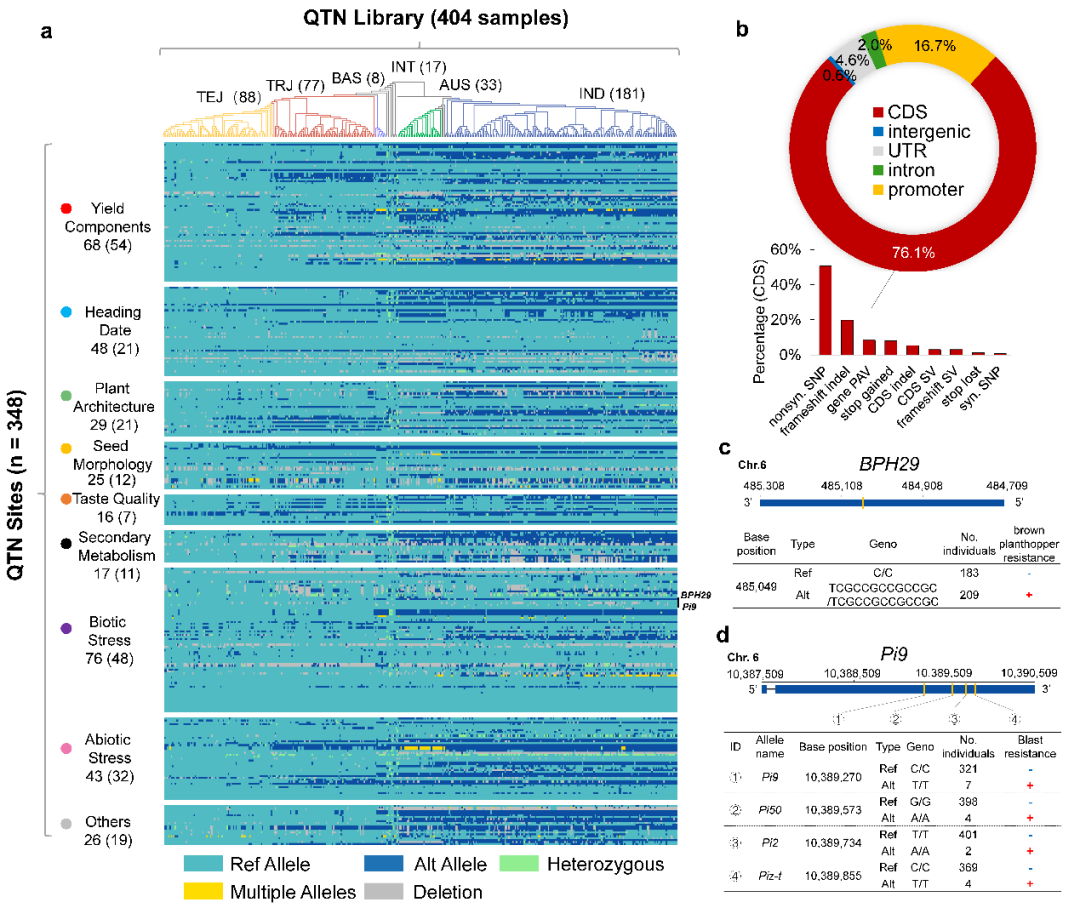
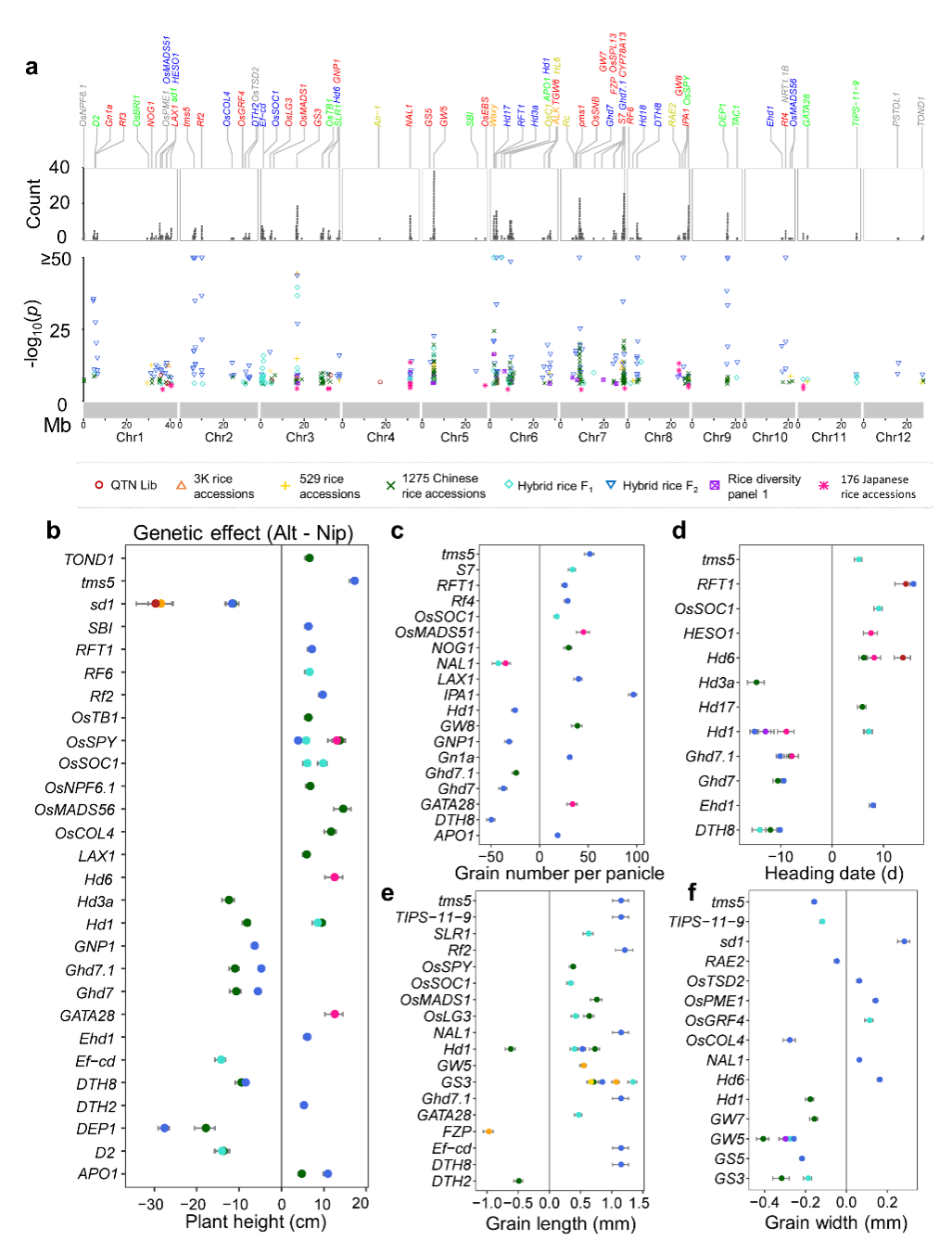
基因变异的效应评估
研究团队选用了八套遗传群体,利用这些群体大规模的原始测序数据和丰富的表型数据,在同一尺度下重新分析,对QTN的效应强弱进行了统一的量化评估,包括同一QTN在不同亚种背景下、在不同地域环境下的遗传效应。结合每个基因的原始文献描述,该图谱为每个QTN提供了精准的效应方向和强弱的数字化注解。
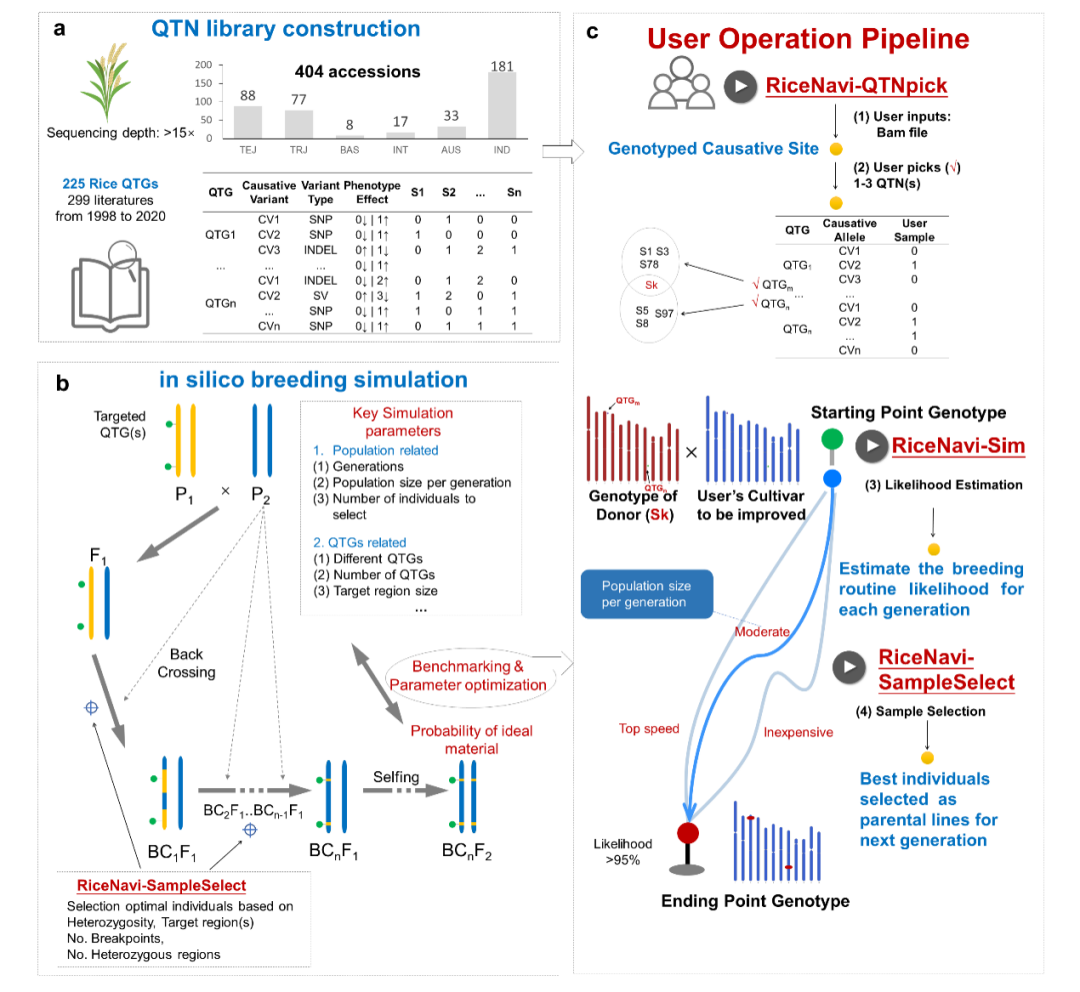
水稻版的“地图导航”程序
常用的地图导航有三个主要功能:一、定位用户位置并展示周边路况;二、到达目的地最优路线规划和时间预估;三、行驶过程中语音提示直行或转弯。对标这三大功能,论文作者开发了一套水稻版的导航软件包。利用水稻QTN图谱和遗传图,论文作者系统分析了水稻基因组中存在的遗传累赘,并针对杂交-回交-自交、群体样本量、导入位点数等各类情形进行了大数据仿真模拟,获得了育种设计路线的优化参数。
研究人员最终开发出了针对普通用户的RiceNavi网站平台和针对服务器用户的离线版RiceNavi软件包,配备了三大功能:一、提供用户待改良品种的全部QTN基因型,并展示实体库中具有互补等位的种质材料;二、根据用户需要导入的基因位点(1-4个)给出最优育种路线;三、育种过程每一代根据中间群体的基因型挑出最有潜力的若干后代材料。
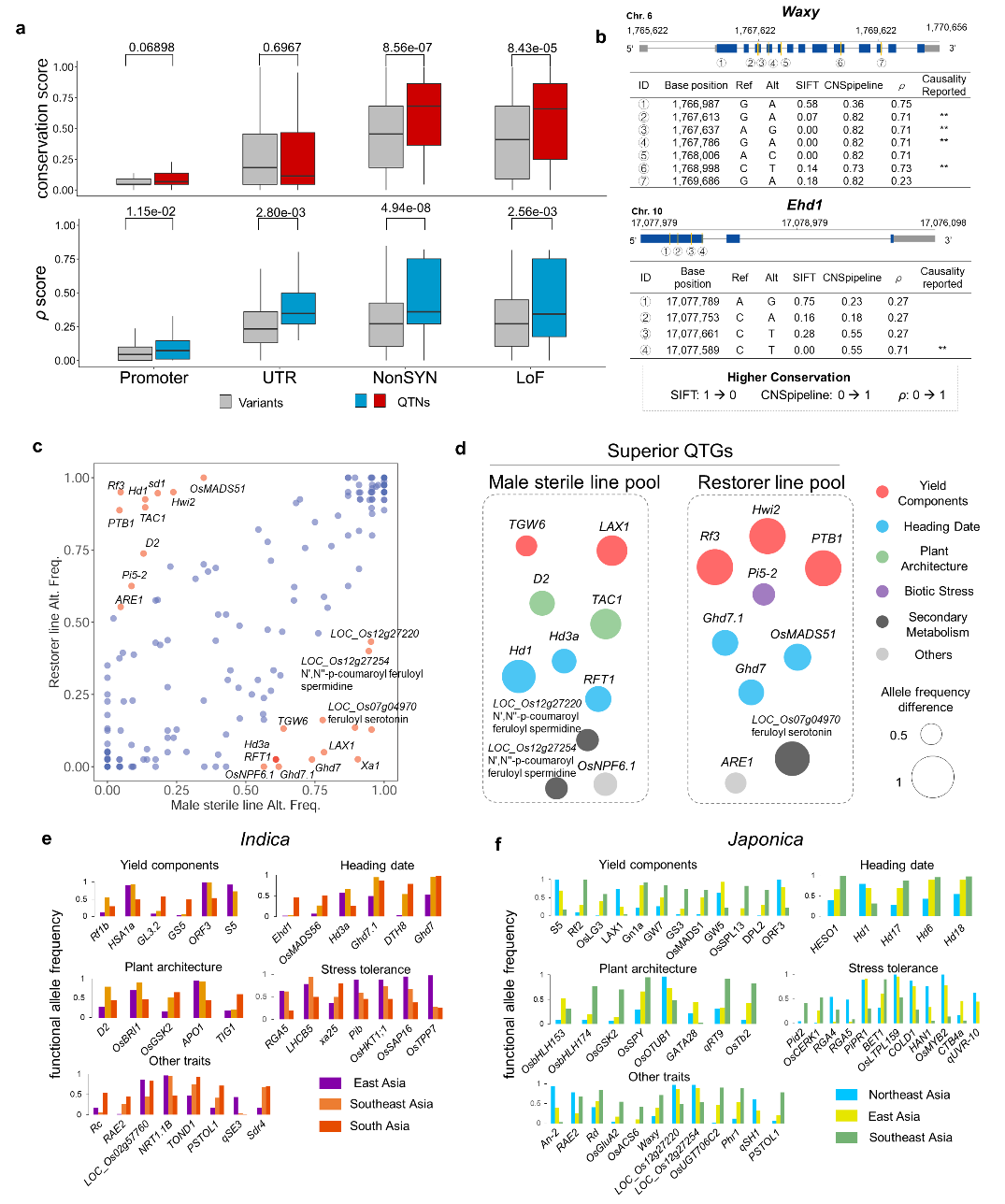
遗传分析上的应用例证
结合不同类型的遗传学问题,RiceNavi可以服务于各类的遗传学分析。
作为示例,论文作者利用典型的粳稻、籼稻群体,鉴定到了决定籼粳分化性状的基因集;
利用野生稻、地方种和育成种群体,揭示出与野生稻驯化、现代育种改良等相关的基因及其变迁趋势;
利用来自不同生态区品种材料,揭示出与地区环境适应性相关的基因及其分布规律;
利用杂交稻不育系群体、恢复系群体,鉴定出与杂种优势、配合力相关的候选基因;
利用这些QTN在基因组中的分布规律,揭示出大尺度进化过程中基因编码和调控区功能元件的保守性,将为大规模组学数据深度学习模型的调试提供帮助。
设计育种上的应用例证
作为示例,RiceNavi还被应用于常规稻主栽品种“黄华占”的改良中。借助于RiceNavi的选配指导和路线优化,仅用两年半时间实现了既定育种目标,获得了株型紧凑、生育期略短、有香味的黄华占。
现阶段,由于QTL基因尚未被全部克隆、遗传互作及遗传-环境互作了解不多,导致分子育种存在盲区,造成有些时候虽然QTN被精准导入,但并不能发挥预期作用。为了掌握这些盲区对分子育种到底影响有多大,该研究构建了271份导入系,其中65.7%材料的性状变化完全符合预期、15.5%部分符合、18.8%不符合。随着水稻遗传学研究的深入,这些盲区有望被系统性地解决,RiceNavi也将不断更新、升级、完善。
上海师范大学魏鑫副研究员和邱杰副研究员为论文共同第一作者,博士研究生雍开成、范炯炯、张绮等参加了材料的鉴定考察和遗传改良,黄学辉教授为通讯作者。
该研究的种质资源材料主要受益于中国农科院等多个课题组的支持。中科院韩斌院士、Jeremy Murray研究员、美国华盛顿大学Kenneth M. Olsen教授、上海师范大学许萍老师和张琳老师等参与部分研究或提供重要帮助。研究得到了国家自然科学基金重大研究计划、国家杰青等项目的资助。
软件链接:
http://www.xhhuanglab.cn/tool/RiceNavi.html
论文链接:
https://www.nature.com/articles/s41588-020-00769-9
扫二维码用手机看
热门推荐
版权所有 © 2021 河南省作物分子育种研究院 豫ICP备16004882号-1 网站建设:中企动力 郑州 营业执照 本网站已支持Ipv6


 0371-65738246
0371-65738246

 在线留言
在线留言 